FAG resymé
Helgenomsekvensering av SARS-CoV-2 gir mer presis smitteoppsporing
En studie på helgenomsekvensering av SARS-CoV-2 i smitteoppsporing blant sykehusansatte, viser at oppsporingen blir mer presis med bruk av virussekvensering.
Av HEGE VANGSTEIN AAMOT, forsker og bioingeniør, PhD, Mikrobiologi og smittevern, Ahus
Helgenomsekvensering er en analysemetode som brukes for å undersøke arvematerialet, genomet, til blant annet bakterier og virus. Metoden er blitt mer utbredt i overvåking og smitteoppsporing i forbindelse med mistenkte utbrudd.
Allerede i begynnelsen av 2020 ble det utviklet en analyseprotokoll for helgenomsekvensering av SARS-CoV-2, som i ettertid har blitt brukt over hele verden for å kunne undersøke spredning og utvikling av pandemiviruset.
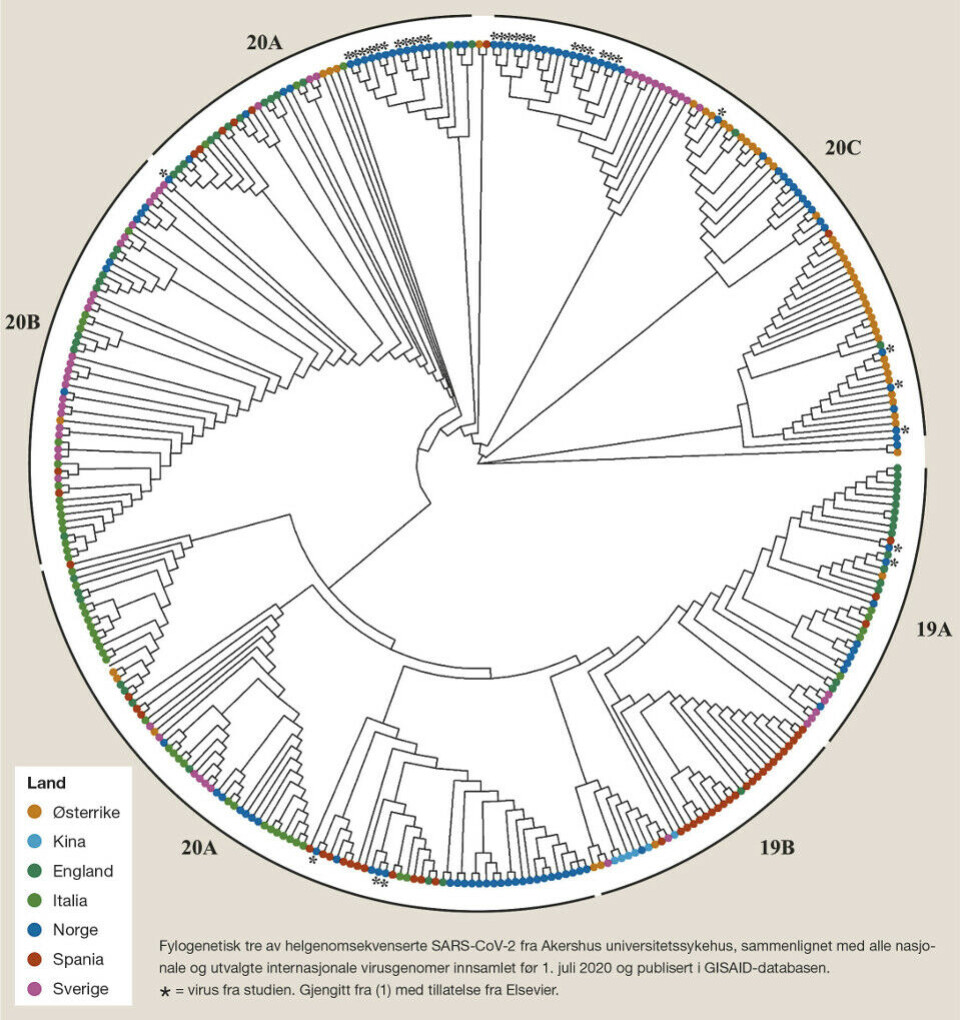
I en nylig publisert artikkel i Journal of Hospital Infection (1), viser forskere ved Akershus universitetssykehus (Ahus) og OsloMet nytteverdien av kombinasjonen av helgenomsekvensering av SARS-CoV-2 og kartlegging av nærkontakter i smitteoppsporing.
Fem mistenkte utbrudd blant ansatte ved Ahus ble undersøkt. To utbrudd ble bekreftet og ett avkreftet. I tillegg ble et mulig utbrudd påvist, som ikke var blitt oppdaget med tradisjonell smittesporing. Ved å undersøke hele virusgenomet oppnås en høyere oppløsning, som gjør det mulig å påvise mutasjoner og endringer i arvematerialet etter hvert som smitten sprer seg. Studien viste at nye mutasjoner kan oppstå mellom smittetilfellene.
Artic Network-protokollen brukt i studien inneholder følgende trinn:
- cDNA syntese av SARS-CoV-2 RNA. SARS-CoV-2 er et av de største RNA-virusene (nesten 30kb).
- PCR amplifisering. Amplifisering av virusgenomet ved hjelp av to overlappende multiplex PCR, som til sammen dekket hele genomet. Det er til sammen 218 primerpar.
- Forbehandling av PCR-produktene før sekvensering. Dette innebærer blant annet merking med barkoder, slik at man kan sekvensere flere prøver samtidig. Typisk sekvenseres 24-48 prøver parallelt. Det er mulig å bruke flere barkoder, men hvis man øker antallet prøver vil man også måtte øke sekvenseringstiden ettersom det vil ta lengre tid å få nok kopier til helgenomanalyse.
- Oppsett for nanoporesekvensering. Det ble brukt nanoporesekvensering i denne studien, fordi det da er mulig å analysere sekvensene i det de blir produsert og man kan kjøre mindre antall prøver. Det finnes også oppsett for Illumina-sekvensering.
- Bioinformatiske analyser. Utføres for å detektere mutasjoner og undersøke slektskap.
Helgenomsekvensering av SARS-CoV-2 er nå en del av diagnostikken ved Ahus. Målet er å sekvensere virus fra alle positive pasienter og ansatte ved sykehuset. Dette gir god oversikt over mulige utbrudd, og kan hjelpe med å forbedre smitteverntiltakene og avdekke mulige brudd i eksisterende smittevernrutiner. I tillegg følger hele verden nøye med på nye virusvarianter, som kan føre til økt smitte, mer alvorlig sykdom og vaksinesvikt.
Studien ble utført av Alexander H. Løvestad (doktorgradsstipendiat, Institutt for naturvitenskapelige helsefag og bioingeniørfag, OsloMet), Silje Bakken Jørgensen (smittevernoverlege, PhD, Mikrobiologi og smittevern, Ahus), Nina Handal (seksjonsoverlege, Mikrobiologi og smittevern, Ahus), Ole Herman Ambur (førsteamanuensis, Institutt for naturvitenskapelige helsefag og bioingeniørfag, OsloMet) og Hege Vangstein Aamot (forsker og bioingeniør, PhD, Mikrobiologi og smittevern, Ahus).
Referanse
- Løvestad AH, Jørgensen SB, Handal N, Ambur OH, Aamot HV. Investigation of intra-hospital SARS-CoV-2 transmission using nanopore whole-genome sequencing. J Hosp Infect. 2021;111:107-16.
























