FAG i praksis
Studenter var involvert i utvikling av nytt molekylærpatologisk laboratoriekurs
Studentene etablerte disse metodene: Real-Time qPCR, fluorescens in situ hybridisering (FISH) og G-bånd-karyotypering. Metodene ble tatt i bruk i undervisningen i patologi høsten 2017.
Studenter var involvert i utvikling av nytt molekylærpatologisk laboratoriekurs.pdf(227 KB)
Innledning
I patologiemnet ved bioingeniørutdanningen, Høgskulen på Vestlandet (HVL), avdeling Bergen, skal studentene lære hvordan sykdommer fører til forandringer i celler og vev. De skal videre kjenne til grunnleggende metoder innen patologisk diagnostikk. Patologi er det eneste medisinske laboratorieemnet hvor utdanningen ved HVL ikke har etablert eget internt laboratoriekurs. Den praktiske laboratorieerfaringen studentene får i dag er via ekstern praksis i histologi og cytologi ved avdeling for patologi, Haukeland Universitetssykehus. Diagnosene innen patologi baserer seg fortsatt i stor grad på tradisjonell makro- og mikroskopisk undersøkelse av pasientmateriale. Senere utvikling innen molekylærpatologi har ført til identifisering av spesifikke sykdomsmarkører i vev og celler, noe som er viktig både for å stille presis diagnose og for persontilpasset behandling. Dermed har molekylærpatologiske analyser av DNA/RNA- og proteinmarkører i større grad blitt tatt i bruk i patologilaboratorier og i diagnostikken av flere sykdommer. Utdanningen har derfor hatt som mål å etablere molekylærpatologiske analyser til et internt laboratoriekurs på HVL.
Med utgangspunkt i en kommersiell leukemicellelinje, Molm-13, har studentene etablert følgende analyser for laboratoriekurset: Real-Time qPCR, fluorescens in situ hybridisering (FISH) og G-bånd-karyotypering. Bachelorstudentene har også utarbeidet en film som viser bruk av Real-Time qPCR-instrumentet. Filmen skal brukes til ePraksis for studentene i laboratoriekurset.
Patologisk kasus
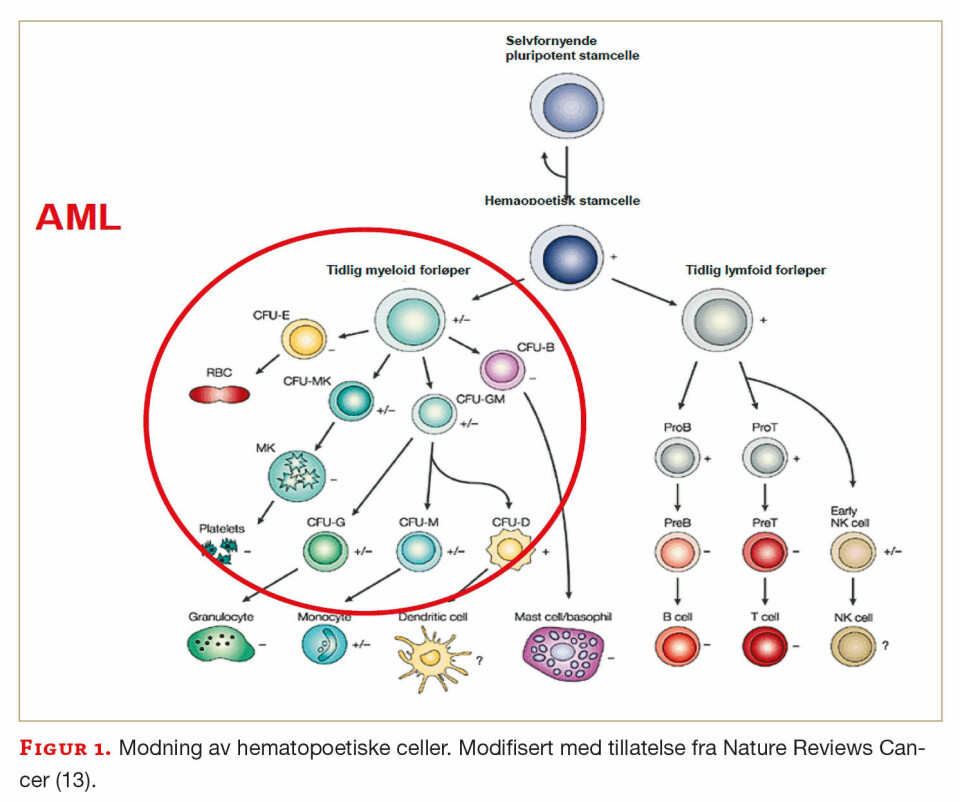
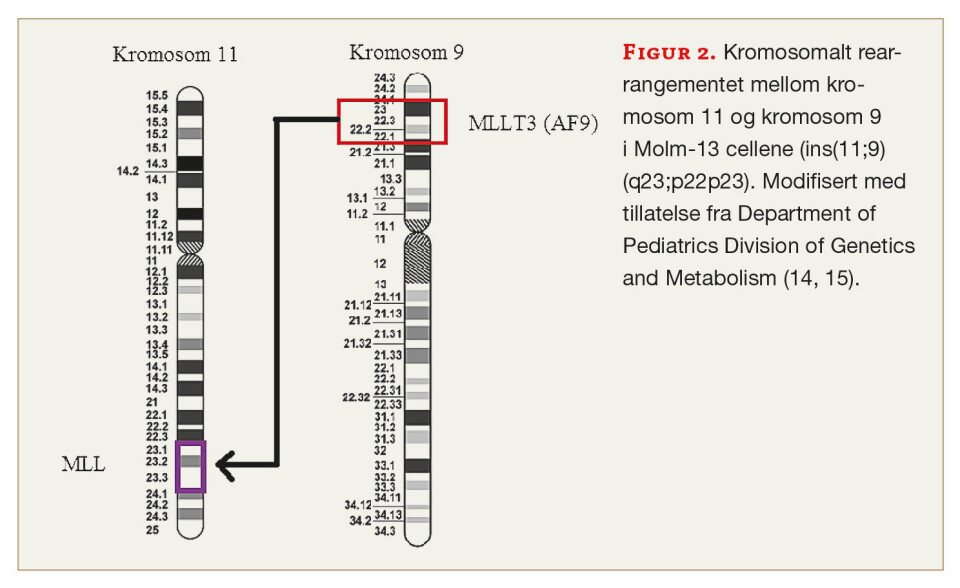
Akutt myelogen leukemi (AML) er en blodkreftsykdom som utvikles fra myeloide celler hvor stamcellene fra beinmargen ikke gjennomgår normal modning (differensiering) til normale hvite blodceller, men resulterer i umodne (abnormale) myeloblaster (figur 1) (1). Molm-13 er en kommersielt tilgjengelig cellelinje isolert fra perifert blod fra en pasient med AML FAB M5a. Dette er en aggressiv subtype av AML som er assosiert med en dårlig prognose (4). Hos denne pasienten er leukemitypen utviklet fra myelodysplastisk syndrom og uttrykker i henhold til litteraturen pentasomi 8 og trisomi 13 (2). Molm-13 innehar en kromosomal insersjon forårsaket av et kromosomalt rearrangement (3). Deler av MLLT3-genet (koder for proteinet AF9) lokalisert på kromosom 9 bånd p22p23, har fusjonert inn i MLL-genet (koder for proteinet Lysin Metyltransferase 2A), som er lokalisert på kromosom 11 bånd q23. De kromosomale endringene (ins(11;9) (q23;p22p23)) er vist i figur 2. Dette rearrangementet gir opphav til et MLL-AF9 onkogen, og ekspresjon av fusjons mRNA (MLL-AF9), som hindrer normal modning av hematopoetiske celler (figur 2). Insersjonen kan påvises både i genomisk DNA (gDNA) og på messenger RNA (mRNA)-nivå. Flytdiagram over analysene utført på Molm-13 cellelinjen for identifisering av patologisk markør, er vist i figur 3.
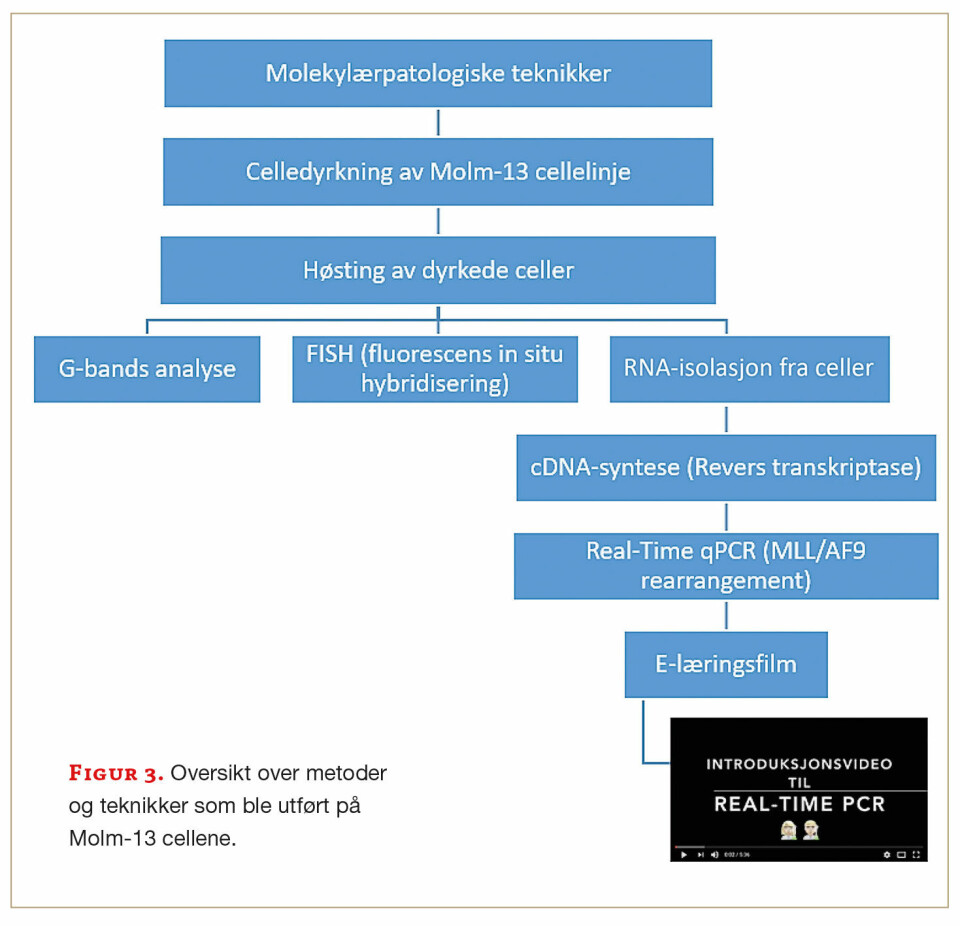
Molekylærpatologiske analyser og funn
Celledyrkning
Den kommersielle cellelinjen Molm-13 (Leibniz Institute DSMZ) ble dyrket i RPMI-medium tilsatt Penicillin-Streptomysin (1 %), L-glutamin (1 %), føtalt bovint serum (10 %) ved 37°C og 5 % CO2 (2).
RNA-isolasjon og cDNA-syntese
RNA ble isolert fra ca. 10 millioner sentrifugerte og pelleterte celler, ved bruk av PureLinkRNA Mini Kit (Invitrogen av Thermo Fisher Scientific, Waltham, MA, USA) i henhold til anbefalinger av produsent (5). Cirka 400 ng total RNA ble benyttet videre i cDNA-syntesen (revers transkriptasereaksjon) ved bruk av enzymkitet SuperScript™ IN VILO™ Master Mix with ezDNase (Invitrogen av Thermo Fisher Scientific) i henhold til protokoll (6).
Real-Time qPCR
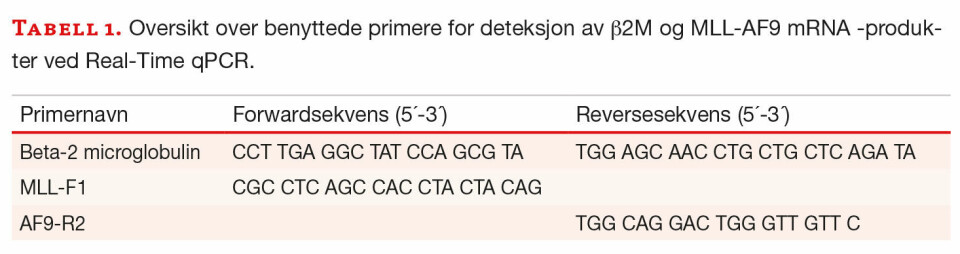
Kvantitativ (q)PCR for påvisning av MLL-AF9 fusjons-mRNA ble utført på StepOnePlus Real-Time PCR system (Applied Biosystems, Foster city, CA, USA). Her ble et Fast-program på 40 min bestående av følgende steg brukt: Holding stage (95 °C i 2 sek) og Cycling stage (95 °C i 1 sek og 60 °C i 20 sek over 40 PCR-sykler). Fast-program ble valgt for å redusere analysetiden i et laboratoriekurs (7). Kitet PowerUp™ SYBR™ Green Master Mix (Applied Biosystems) (8), bestående av SYBR Green dye, Taq polymerase, dNTP, ROX passiv referansefarge og optimaliserte bufferkomponenter, ble blandet med nysyntetisert cDNA og primere spesifikke for amplifisering av MLL-AF9 fusjonsprodukt og et egnet referansegen. Titrering av mengder cDNA og PowerUp™ SYBR™ Green Master Mix for optimalt resultat, ble utført i henhold til anbefalinger fra produsenten (9). Primere brukt til amplifisering av MLL-AF9-produktet og referansegenet beta-2-mikroglobulin (β2M), er spesifisert i tabell 1. En økning i fluorescensintensitet ved Real-time qPCR vil være et mål på mengden av MLL-AF9 eller β2M PCR-produkt. PCR-produkt detekteres når fluorescensintensiteten, det vil si mengden av produkt, stiger over forventet fluorescens bakgrunnsnivå ved cycle of threshold (ct-verdi) (7). En negativ kontroll (blankprøve som inneholder vann i stedet for cDNA templat) ble inkludert for å avkrefte uspesifikt PCR-produkt produsert i reaksjonen.
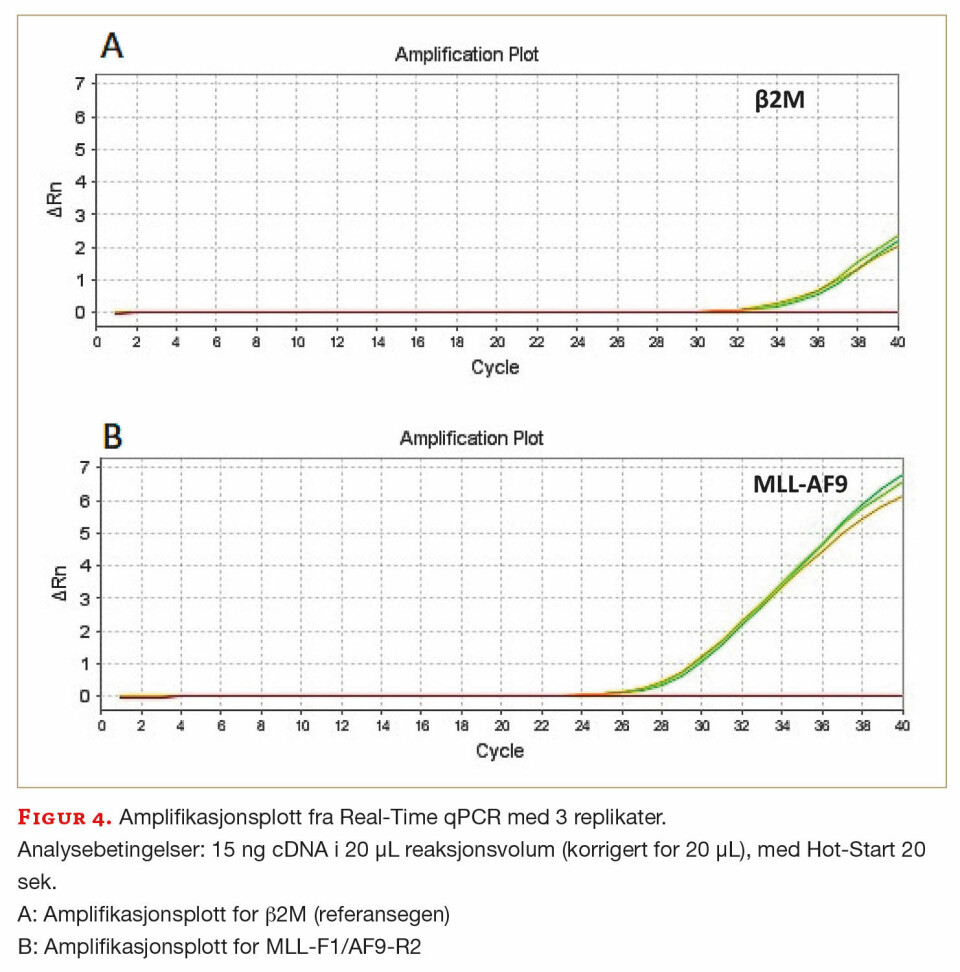
Tilstedeværelsen og mengden β2M og MLL-AF9 presenteres i figur 4 i sine respektive amplifikasjonsplot. ΔRn på y-aksen viser intensitet av fluorescenssignalet til PCR-produktet for β2M (4A) og MLL-AF9 (4B), og er plottet mot antall PCR-sykler på x-aksen. Etter optimalisering av reaksjonsmengder fant vi at ca. 15 ng cDNA i 20 µl reaksjonsvolum gav mest optimalt resultat for amplifikasjon av det interne referansegenet β2M, med grei signalintensitet og basert på tre jevne paralleller (grønne kurver i figur 4A). For å ytterligere øke fluorescensintensiteten på kurvene (øke mengden amplifisert produkt) kan en øke antall PCR sykler i fast-programmet. Ved bruk av primersett MLL-F1/AF9-R2 (tabell 1) ble MLL-AF9-fusjonsproduktet best påvist også ved 15 ng cDNA og 20 µl totalvolum i reaksjonen (figur 4B). Blankprøven for begge qPCR-oppsett var negative for produkt, noe som indikerer ingen forurensning i reaksjonsmiksen (rød kurve i figur 4A og 4B).
FISH
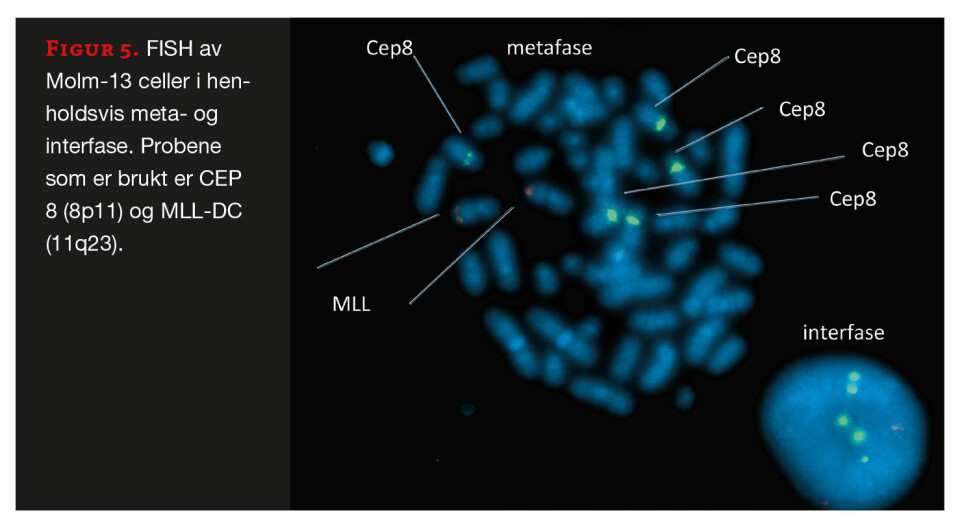
Fluorescens in situ hybridisering (FISH) er en molekylærbiologisk teknikk hvor man benytter fluorescensmerkede prober for spesifikk merking av hele eller deler av et kromosom. Teknikken benyttes blant annet til å identifisere genetiske forandringer som for eksempel tap av kromosomalt materiale (delesjoner), tillegg av kromosomalt materiale (duplikasjoner), forbytting av kromosomalt materiale mellom to kromosomer (translokasjoner) eller innsetting av kromosomalt materiale (insersjoner). Hvilken FISH-probe man velger er basert på hvilket område i gDNA, på et bestemt kromosom, man ønsker å studere (10). Det finnes ulike typer fluorescerende prober, deriblant locusspesifikke prober (binder til et spesifikt område på et kromosom), sentromerprober (hybridiserer til sekvenser i midten av et kromosom, sentromerområdet) og paint prober (farger hele kromosomer) (11). Våre kromosomanalyser ble utført i metafasen. For dette ble Molm-13 cellene forbehandlet med colcemid, før cellene ble merket med en locuspesifikk probe som binder til MLL-genet (MLL-DC-split probe; locus 11q23). Det ble i tillegg benyttet en sentromer-spesifikk probe mot kromosom 8 (CEP8; locus 8p11). Begge probene ble behandlet i henhold til anbefalinger fra leverandør (Abbott Laboratories, Abbott Park, IL, USA). MLL-proben ble valgt på grunnlag av informasjon om rearrangement i dette genet, og fordi det er en av probene som diagnostisk blir benyttet ved mistanke om AML for prognostisk kartlegging. CEP8-proben ble valgt etter informasjon om tilstedeværelse av flere kromosom 8 hos pasienten som Molm-13 cellene stammet fra (1). FISH-analysene i Molm-13-cellene viste tilstedeværelse av fem kopier av kromosom 8 ved merking med CEP8-proben (figur 5). Det ble observert to normale signaler på kromosom 11 med MLL-DC-split-proben. Dette er som forventet da det tidligere er blitt vist at dette MLL-rearrangementet med insersjon ikke kan detekteres ved hjelp av MLL FISH-probe (3) i motsetning til MLL-rearrangementer basert på kromosomale translokasjoner.
Vi konkluderte derfor med at FISH-analyse for deteksjon av MLL-rearrangementet, slik det foreligger i Molm-13 cellene, er en mindre egnet analyse enn Real-time qPCR.
G-bånd-karyotypering
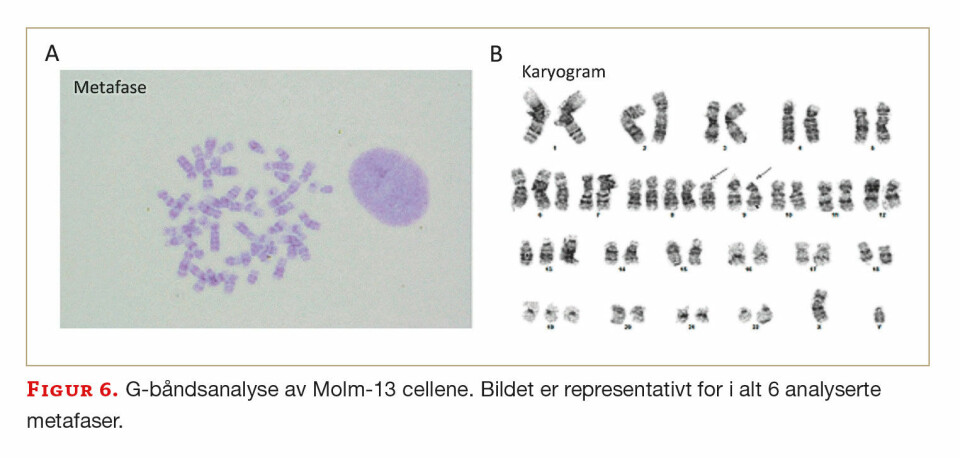
Dette er en undersøkelse som utføres på kromosomer etter dyrking og høsting av celler. Ved analysen ble seks Molm-13-celler i metafasestadiet analysert for kromosomfeil. Dette ble gjort ved at cellene, etter høsting, ble behandlet med trypsin for å «åpne» DNA-strukturen og gjøre den tilgjengelig for påfølgende Leishman-farge (Merck, Kenilworth, NJ, USA), som farger genfattige og genrike områder i forskjellige fargenyanser. Resultatet, som er vist i figur 6, ble analysert med et Nicon eclipse Ci mikroskop og fotografert i Brightfield (svart/hvitt lys) på et fluorescensmikroskop. En metafase ble karyotypert med det avanserte bildeanalyseprogrammet Cytovision (Leica Biosystems, Nussloch, Tyskland). Seks metafaser ble analysert for å sikre at kromosomavvikene var gjennomgående i cellelinjen. Den endelige karyotypen som ble ferdigstilt i Molm-13-cellene da alle analyser var utført (FISH og Real-Time qPCR, supplert med G-bånd-karyotypering), indikerte følgende kromosomavvik: 52, XY, +6, +8, +8, +8, ins(11;9) (q23;p22p23), +13,+19, noe som bekreftet pentasomi 8 og trisomi 13, som også er i tråd med hva som er oppgitt i litteraturen (3). I det molekylærpatologiske laboratoriekurset vil studentene øve seg på karyotypering i XY-karyotype-programvaren (Learn genetics, University of Utah) (12) på normale kromosomer.
ePraksis
Det ble i sammenheng med Real-Time qPCR oppsettet på Molm-13 cellene laget en film som kan benyttes i ePraksis på laboratoriekurset i molekylærpatologi. Filmen tar for seg prosessen fra prøvepreparering til tolkning av resultat på StepOnePlus Real-Time PCR instrumentet. Det endelige produktet, som er egenhendig produsert av bachelorstudentene i løpet av prosjektperioden, er tilgjengelig via følgende link: https://www.youtube.com/watch?v=QFQTybdqBmE
Konklusjon
G-båndsanalyse og FISH-analyse bekreftet pentasomi 8 i Molm-13 cellene. FISH-analyse kan derimot ikke benyttes til sikker påvisning av MLL rearrangementet i Molm-13 cellene ved bruk av den valgte MLL-DC-split-proben. FISH og G-bånd-karyotypering kan imidlertid tas i bruk på laboratoriekurset, for å sikre forståelsen av tilgjengelige metoder i leukemidiagnostikk, og begrunne bruken av Real-Time qPCR som ideell metode i påvisning av MLL rearrangementet og ekspresjon av MLL-AF9 fusjons mRNA i Molm-13 cellene. Denne kombinasjonen av metoder ble vurdert som mest optimal, og supplert med ePraksis-filmen, ble de inkludert i utdanningen høsten 2017.
Takk
Takk til Helle Lybæk, forsker ved Senter for medisinsk genetikk og molekylærmedisin, Haukeland Universitetssykehus, for kritisk gjennomlesning av manuskriptet og nyttige kommentarer.
Også takk til Hilde Mæland ved Senter for medisinsk genetikk og molekylærmedisin for hjelp til karyotyping av tilsendte metafaser.
Forfatterne oppgir ingen interessekonflikter.
Artikkelen bygger på en bacheloroppgave fra 2017 utført ved bioingeniørutdanningen.
























